COVID-19 - Trajetória de Crescimento dos Estados do Brasil
Introdução
Recentemente, tive contato com o projeto do Observatório COVID-19 BR, cujo link é https://covid19br.github.io/. O site organiza uma base de dados informada pelo Ministério da Saúde que nos permite obter informações dos Estados e dos Municípios do Brasil.
O nosso interesse aqui é algo relativamente simples. Tendo o conhecimento dessa base de dados, queremos filtrar os dados dos Estados e plotar os pontos e tendência de novos casos confirmados em log, para inferir se a taxa de crescimento está acelerando, estável ou desacelerando.
Obtenção dos Dados
O passo inicial para a obtençao dos dados é definir o link para o download, o formato do arquivo e se será necessária a descompressão. No nosso caso, os dados disponibilizados são zipados na extensão “.gz”. Após de-zipados, temos a extensão “.csv”. O acesso para download é através do link: https://data.brasil.io/dataset/covid19/caso_full.csv.gz.
Dessa forma, começamos limpando o global environment com o comando rm(list=ls()). Carregamos os seguintes pacotes necessários: R.utils para de-zipar o arquivo “.gz”, readr para ler o formato “.csv”, dplyr para manipular a base de dados, scales para poder alterar o eixo y do gráfico para log e ggplot2 para plotar os gráficos.
Na sequência, fazemos o seguinte: (1) definimos o link para download; (2) Definimos um arquivo temporário com a extensão “.gz” para o download; (3) Fazemos o download usando a URL e o arquivo temporário como destino; (4) Removemos algum arquivo anterior que lá esteja de-zipado; (5) De-zipamos o arquivo para obter apenas o arquivo “.csv”.
rm(list=ls())
library(R.utils)
library(readr)
library(dplyr)
library(scales)
library(ggplot2)
URL <- "https://data.brasil.io/dataset/covid19/caso_full.csv.gz"
Temp <- tempfile(fileext = ".gz")
download.file(URL, destfile = Temp, mode = 'wb')
file.remove("./Aprendizado/COVID-19/Micro.csv")## [1] TRUEgunzip(Temp, destname = "./Aprendizado/COVID-19/Micro.csv")Dados obtidos, temos agora que importar o “.csv” para um data frame. Depois de importado, podemos usar o comando head para visualizar as primeiras informações da tabela.
Micro <- read_csv("Aprendizado/COVID-19/Micro.csv")
head(Micro, 10)## # A tibble: 10 x 15
## city city_ibge_code date estimated_popul~ is_repeated is_last
## <chr> <dbl> <date> <dbl> <lgl> <lgl>
## 1 São ~ 3550308 2020-02-25 12252023 FALSE FALSE
## 2 <NA> 35 2020-02-25 45919049 FALSE FALSE
## 3 São ~ 3550308 2020-02-26 12252023 FALSE FALSE
## 4 <NA> 35 2020-02-26 45919049 FALSE FALSE
## 5 São ~ 3550308 2020-02-27 12252023 FALSE FALSE
## 6 <NA> 35 2020-02-27 45919049 FALSE FALSE
## 7 São ~ 3550308 2020-02-28 12252023 FALSE FALSE
## 8 <NA> 35 2020-02-28 45919049 FALSE FALSE
## 9 São ~ 3550308 2020-02-29 12252023 FALSE FALSE
## 10 <NA> 35 2020-02-29 45919049 FALSE FALSE
## # ... with 9 more variables: last_available_confirmed <dbl>,
## # last_available_confirmed_per_100k_inhabitants <dbl>,
## # last_available_date <date>, last_available_death_rate <dbl>,
## # last_available_deaths <dbl>, place_type <chr>, state <chr>,
## # new_confirmed <dbl>, new_deaths <dbl>Definido o data frame, temos agora que manipulá-lo para filtrar e organizar as variáveis de interesse. Nisso, o pacote dplyr será muito útil. Usamos a função filter() para filtrar apenas os estados e excluir os municípios. A função group_by() nos permite agrupar as observações de estados por datas e ao fim a função select() nos permite selecionar apenas as colunas da tabela de nosso interesse.
Micro <- read_csv("Aprendizado/COVID-19/Micro.csv")
Estados_DF <- Micro %>%
filter(place_type == "state") %>%
group_by(state, date) %>%
select(date, state, new_confirmed)
head(Estados_DF)## # A tibble: 6 x 3
## # Groups: state, date [6]
## date state new_confirmed
## <date> <chr> <dbl>
## 1 2020-02-25 SP 0
## 2 2020-02-26 SP 0
## 3 2020-02-27 SP 0
## 4 2020-02-28 SP 1
## 5 2020-02-29 SP 0
## 6 2020-03-01 SP 0Gráficos das Séries
Tendo sido os dados tratados, vamos agora utilizar o pacote ggplot2. Primeiro definimos o fundo branco a partir da troca de tema, via função: theme_set(theme_bw()). Na sequência, abrimos o plot via função ggplot(), pedimos o plot dos pontos diários, separando os estados por cor através da função geom_point(). Definimos os textos de título e eixos via xlab(),ylab() e ggtitle(). Isso posto, pedimos para gerar uma reta de tendência através do método de “LOESS” que é uma regressão local que permite formato polinomial. A função scale_y_continuous() permite mudar o eixo y para o formato log e facet_wrap() plotar em distintas “facetas”.
theme_set(theme_bw())
ggplot(data = Estados_DF, aes(y = new_confirmed, x = date)) +
geom_point(aes(color = state)) +
xlab('Tempo (dias)') +
ylab('Novos Casos') +
ggtitle('Novos Casos - Covid-19 - Estados BR') +
geom_smooth(aes(fill = state), col = "black",
method = "loess", se = T) +
scale_y_continuous(trans = log2_trans()) +
facet_wrap( ~ state) +
scale_x_date(date_breaks = "months" , date_labels = "%b")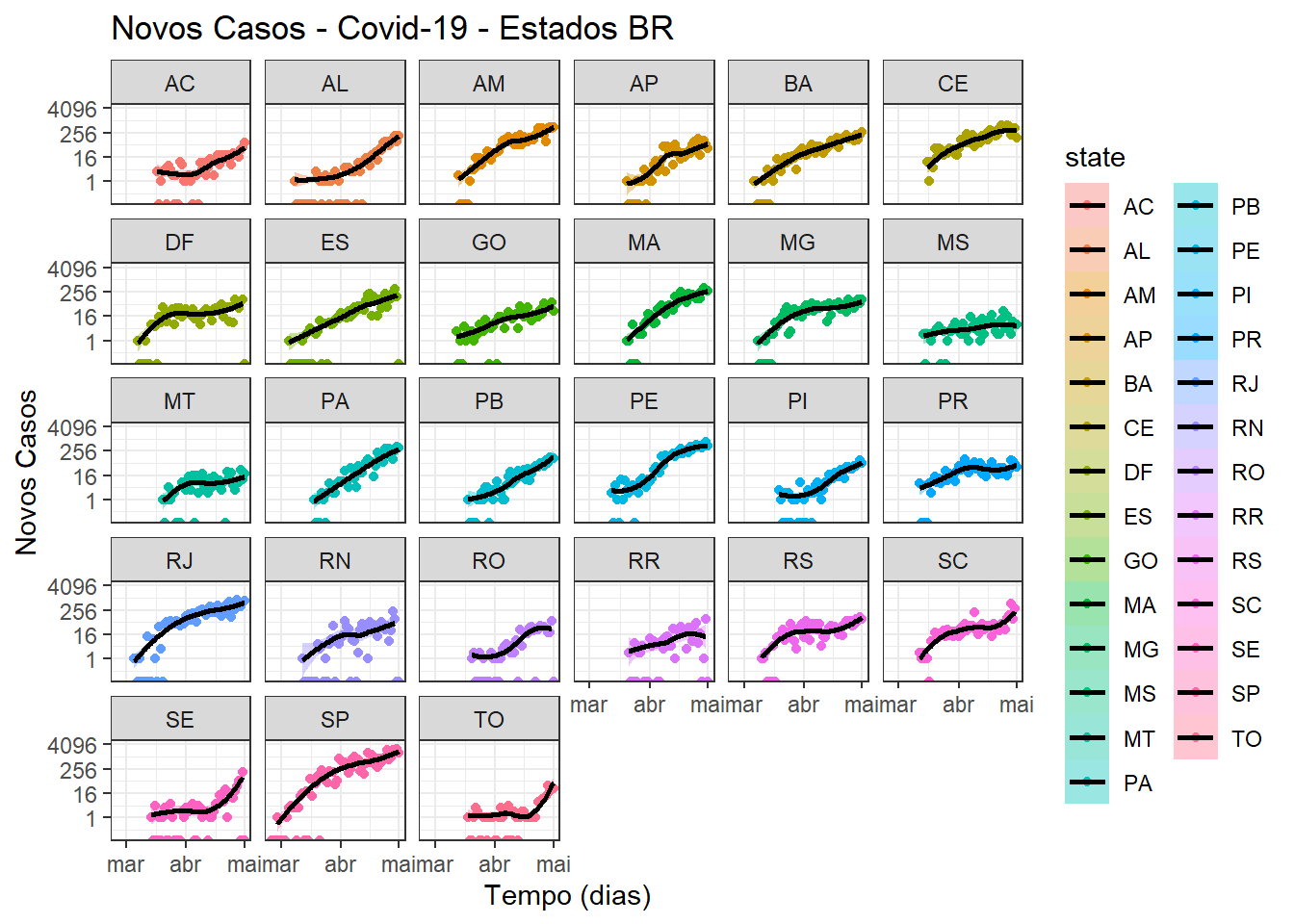
Tendo o gráfico acima, com as devidas retas de tendência estimadas, podemos verificar que os Estados estão em distintos padrões de crescimento.
Podemos separar em:
- Taxas Crescentes: AC, AL, AP, DF, RS, SC, SE, TO.
- Taxas Constantes (ou perto disso): AL, GO, MT, PA, PB, PI, RN.
- Taxas Decrescentes: BA, CE, ES, MA, MG, MS, PE, RJ, RO, RR, SP.
Dessa forma, nesse momento, parece que as medidas de isolamento devem ser tratadas de maneira distinta (em grau de rigidez) para as distintas UFs.